生物基因體以 DNA 的形式攜帶遺傳訊息,生物會選擇性地擷取特定 DNA 片段,將其轉錄成 RNA,再將 RNA 轉譯成蛋白質。而具有生理功能的蛋白質則在生物體中進行著各式各樣的反應,維持生物體的生理機能。由此可知,蛋白質對於生物體的重要性。
但是大家是否知道,在如同茫茫大海的基因體中,蛋白質是如何從 RNA 中被定義出來的呢?答案是:生物資訊學家在訊息 RNA 上尋找起始密碼子 AUG 與其相互距離最遠的終止密碼子(UAG、 UAA、UGA),來定義蛋白質的轉譯區間,且此轉譯區間必須大於一百個胺基酸,而且區間之間不可以相互重疊(圖一A)。
然而,生物體是否真的按照科學家所定義之區間產生典型的蛋白質呢?亦或是如同訊息 RNA 一樣會有選擇性轉錄作用的發生?另外,起始密碼子一定會是 AUG 嗎?如果真的有跟目前定義不同的非典型蛋白質產生,它們是否具有生理功能與意義呢?關於蛋白質的定義,還有許許多多的疑惑困擾著科學家們,今天就讓我們一起徜徉在基因體海洋中,揭開蛋白質的神秘面紗吧!
藏寶圖:利用核醣體定序分析技術找出轉譯起始點
想要在汪洋大海中尋寶,若能有藏寶圖想必能事半功倍。
在 2009 年以來,Ingolia 與 Gao 博士為主的科學家們,建立核醣體定序分析技術,利用「lactimidomycin(LTM)轉譯抑制劑」,抑制轉譯作用在剛起始的階段,讓核醣體累積在訊息 RNA 上的轉譯起始位置,再搭配「puromycin(PMY)轉譯抑制劑」,將已經進入轉譯延長階段的核醣體解開,讓核醣體只能佔據在轉譯起始位置,最後再透過 RNA 定序找出核醣體所佔據的轉譯起始序列(圖一B)。「cycloheximide(CHX)」則為另一種轉錄抑制劑,可以將轉譯作用抑制在剛起始及已經進入轉譯延長階段的核醣體,可以幫助我們定義出整體的轉譯區間(圖一C)。經由 LTM 與 CHX 兩者訊號比較,可以讓我們更準確的定義訊息 RNA 上的轉譯起始點與轉譯區間。
透過核醣體定序分析技術,科學家們發現,在哺乳類動物細胞中,有許多轉譯起始點並不位於典型定義的起始位置,而且,除了起始於 AUG 密碼子之外,也會起始於跟 AUG 相差一個核酸的密碼子,像是 CUG、ACG 等等。此外,轉譯起始點的位置,不只會位於傳統認知的編碼區,還會位於 5 端與 3 端非編碼區。
最重要的是,這些非典型起始位置所轉譯的蛋白質,在生物體中確實會表現出來,並且具有生理功能。例如:促進癌症細胞生長的「FGF2(fibroblast growth factor2)基因」與抑制癌症細胞生長的「c-myc 基因」,皆具有一個位在 5 端非編碼區的 CUG 非典型轉譯起始點,並且會轉譯出與典型蛋白質功能相異的蛋白質異構體。
有趣的是,外在環境的刺激會改變轉譯起始位置,進而轉譯出功能相異的蛋白質。以「MRPL18 基因」為例,當細胞處在正常環境時,MRPL18 會使用典型 AUG 起始點轉譯蛋白質,並且蛋白質會被傳送到粒腺體內組成核醣體,進而幫助蛋白質的合成。但是,若細胞處在高溫逆境中,MRPL18 基因則會使用下游非典型轉譯起始點 CUG 轉譯蛋白質,並將此非典型轉譯蛋白質送至細胞質組成特殊形態的核醣體,用來幫助轉譯 HSP70 熱休克蛋白,進而幫助細胞維持體內環境的穩定並抵抗高溫逆境。
綜合以上的結果,有了核醣體定序分析技術這張藏寶圖,我們發現了一片有別以往認知的新大陸,對於生物體的生理機制與基因轉譯的調控,有了更進一步的認識。
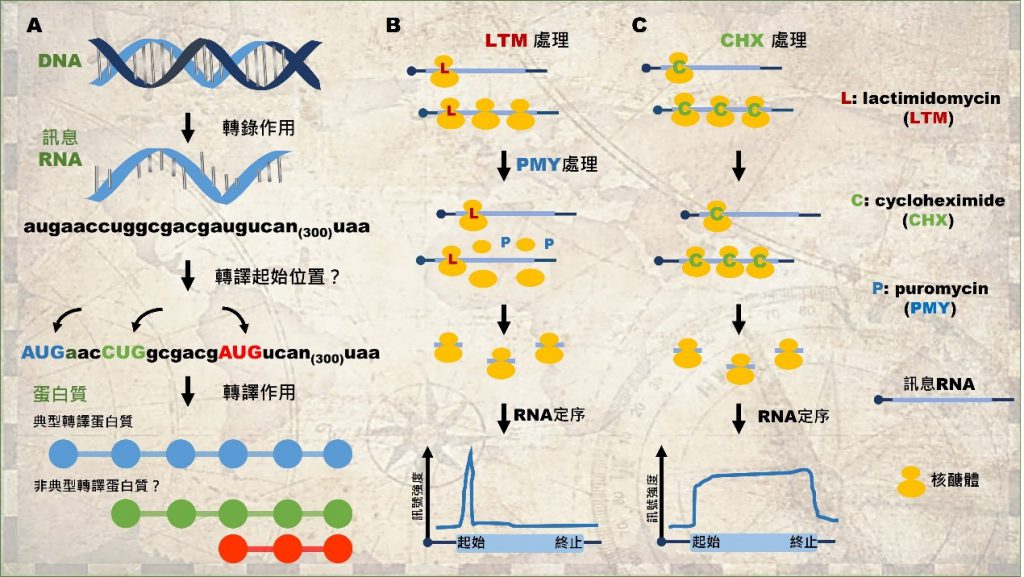
▲圖一:核醣體定序分析技術,幫助定位轉譯起始點。
植物航道中的大秘寶
由於植物無法移動的特性,因此,相對於動物而言,植物更需要透過調控自身蛋白質表現,來因應外在環境的刺激,用以維持生理的穩定。
劉明容博士實驗室於 2020 年透過核醣體定序分析技術,在番茄與阿拉伯芥中,發現超過 50% 的轉譯區間起始於非典型定義位置。如同動物一樣,植物轉譯起始密碼子,可以起始於與 AUG 相差一個核酸的密碼子,例如 CUG 與 ACG,並且發現轉譯起始位置分布於 RNA 的各個位置(5 端非轉譯區、轉譯區、3 端非轉譯區)。另外也發現在有些訊息 RNA 上,透過不同轉譯起始點所產生的蛋白質,表現於不同位置。這樣的研究結果可能代表著,不同轉譯起始點所轉譯的蛋白質,具有不同的生理功能。
另一方面,對於植物而言,植物病毒的入侵將嚴重地影響到作物的產量與質量。因此,若能完整地定義出植物病毒的蛋白質,將有助於植物在抵抗病毒上的應用與發展。以「番茄黃化捲葉病毒(TYLCTHV, genus Begomovirus)」為例,其為單股 DNA 病毒,在其 DNA-A 及 DNA-B 基因體上,各具有六個(AV1、AV2、AC1-AC4)及兩個(BV1、BC1)原始定義基因。這些基因所轉譯出來的蛋白質,在病毒感染植物宿主的過程中,扮演著重要的角色。
透過核醣體定序分析,研究團隊更進一步發現額外 17 個起始於 AUG 與非 AUG 的非典型轉譯起始點,並發現一直以來,對於 AV2 與 BV1 基因的轉譯起始位置,定義是錯誤的,將原始定義之轉譯起始點突變,並不會影響到病毒的致病性;反之,若將新找到的非典型轉譯起始點突變,會抑制 AV2 與 BV1 蛋白質表現,進而降低病毒的致病性。
此外,研究團隊也找到一個尚未被發現的非典型轉譯起始點,其可轉譯出 BV2 蛋白質,並且轉譯區間被隱藏在 BV1 的轉譯區間內,將此非典型轉譯起始點突變會影響到病毒的致病性。基於 BV2 蛋白質分布在細胞的內質網與原生質絲,而內質網與原生質絲是病毒複製及病毒粒在細胞間傳播的必經之地,因而推測 BV2 蛋白質可能參與在這兩項病毒的生理過程,進而幫助病毒在宿主中,完成完整的生命週期。
綜合以上結果,我們可以得知藏寶圖也適用於植物與植物病毒這兩個領域,用來探索非典型轉譯蛋白質的位置。此外,植物與植物病毒的非典型轉譯蛋白質皆具有生理功能。由此可見,生物體藉此增加蛋白質的調控與組成的多樣性,用來維持體內的恆定,抵抗外在的環境刺激。
寶藏的由來?
透過核醣體定序分析技術,我們找到了許多非典型轉譯蛋白。但是,關於生物體如何辨認、決定、甚至如何調控轉譯起始點,至今仍不清楚。若能更進一步地釐清轉譯起始點的調控機制,將有助於瞭解生物體內的運作情形,進而提升生物體抵抗外界刺激,維持體內恆定的能力。
目前針對辨認轉譯起始點的研究方向,主要著重在特定的 RNA 序列與轉譯起始複合體(preinitiation complex;PIC)兩大主軸(圖二)。在特定的 RNA 序列上,Kozak 博士提出的 Kozak 序列,指出在起始位置前方的第三個核酸是 A 或 G,而後方第四個位置是 G,將會有效地提升核醣體與轉譯起始點的結合,進而提升轉譯效率。此外,在動物細胞中也發現,一連串的 GGGGCC 重複性序列會造成核醣體直接進行轉譯作用,產生非典型轉譯蛋白質,並發現其與生物體神經方面的疾病有關,像是一般較為人熟知的漸凍人症狀等等。
另一方面,轉譯起始複合體被發現參與在轉譯起始點的辨認上。其中,由 eIF4A、eIF4E、eIF4G 次單元所組成的 eIF4F 複合體,負責結合到要進行轉譯的 RNA 上,然後招來由 eIF1、eIF1A、eIF2、eIF3、eIF5 次單元所組成的轉譯起始複合體,在 RNA 序列上進行掃描與辨認起始密碼子,進而進行蛋白質的轉譯(圖二)。有研究指出,大量表現 eIF1 次單元可以有效地提升由 AUG 起始密碼子所進行的轉譯作用,而與 eIF2 次單元相似的 eIF2A 與 eIF2D 次單元,則參與在由非 AUG 起始密碼子所進行的轉譯作用中。
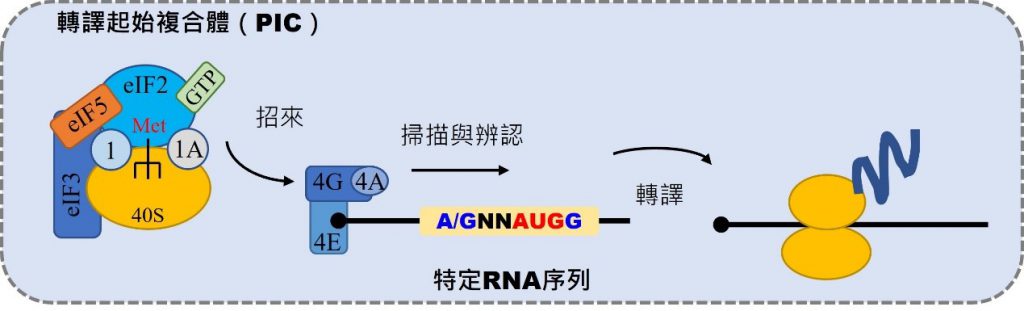
▲圖二:調控轉譯起始點的兩大方向:特定 RNA 序列與轉譯起始複合體。
神秘面紗只揭開了一角
關於轉譯起始點的調控機制,其實尚未釐清,在特定的 RNA 序列上,除了 Kozak 序列外,是否還有其他特殊的序列與轉譯起始點的辨識有關,或是否具有專屬於特定轉譯起始密碼子的 RNA 序列,抑或是,是否具有專屬於特定環境刺激所辨認的 RNA 序列,這些疑問都尚待進一步的分析。在轉譯起始複合體的部分,在不同的環境刺激中,是否會引起不同的次單元表現,進而影響轉譯起始點的選擇呢?
另外,轉譯起始複合體中的次單元究竟是如何影響到轉譯起始點的選擇?除了上述所提到會影響到轉譯起始點選擇的次單元外,其他次單元是否也參與其中?而非典型轉譯蛋白質在各種生理反應中又扮演著什麼樣的角色呢?在探索非典型轉譯蛋白質的功能上,我們任重道遠。
期望未來能破解上述問題,得到的答案應可促使我們更加清楚生物體如何調控蛋白質的轉譯,以及對於現存許多棘手的農作物逆境困境及病害上,提供更好的解藥。除此之外,目前關於非典型轉譯蛋白質的研究,大多集中在哺乳類動物與酵母菌中,在植物及其他農作物是否利用相同的調控機制,或是具有獨特的調控路徑都值得我們去探討,尤其是在糧食作物上,其將有助於幫助植物抵抗目前多變的環境刺激,進而解決糧食危機。
延伸閱讀
1.Gao, X., Wan, J., Liu, B., Ma, M., Shen, B., and Qian, S.B. (2015). Quantitative profiling of initiating ribosomes in vivo. Nat Methods 12, 147-153. 10.1038/nmeth.3208.
2. Li, Y.R., and Liu, M.J. (2020). Prevalence of alternative AUG and non-AUG translation initiators and their regulatory effects across plants. Genome Res 30, 1418-1433. 10.1101/gr.261834.120.
3. Chiu, C.W., Li, Y.R., Lin, C.Y., Yeh, H.H., and Liu, M.J. (2022). Translation initiation landscape profiling reveals hidden open-reading frames required for the pathogenesis of tomato yellow leaf curl Thailand virus. Plant Cell 34, 1804-1821. 10.1093/plcell/koac019.
4. Kearse, M.G., and Wilusz, J.E. (2017). Non-AUG translation: a new start for protein synthesis in eukaryotes. Genes Dev 31, 1717-1731. 10.1101/gad.305250.117.
