蛋白設計在新型功能酵素蛋白及具有醫療潛力的新型蛋白設計近幾年在更強大電腦計算能力及方法學的進步下,不斷的在蛋白設計與實驗印證取得更吻合的例證。自然界中發現共22種胺基酸,其中包含第21胺基酸硒半胱胺酸(selenocysteine)及第22胺基酸吡咯賴胺酸(pyrrolysine)。
在科學界瞭解吡咯賴胺酸體內嵌入的機轉後,吡咯賴胺酸進一步蛋白質工程所能體內嵌入超過200種非典型胺基酸1-3在蛋白設計上已成為新的工具;非典型胺基酸在蛋白序列中造成的蛋白結構變化和影響及新功能蛋白的開發,都是在經過二十年研究讓非典型胺基酸體內嵌入技術更有效率之後的發展趨勢。此文旨在簡介非典型胺基酸體內嵌入研究領域的最新發展與未來展望。
基因密碼轉譯在轉核醣核酸(tRNA)功能確認由三個核酸基因子密碼轉譯為單一胺基酸(amino acid)後,自然系統解碼方式接著被一一確認,在由ATGC四個核酸基因子排列組合所形成64(4×4×4)種組合,其中由61個密碼組合解碼20個天然胺基酸,剩餘TAG,TAA及TGA為停止密碼(stop codon)。繼起始基因密碼ATG訊號開始依信使核醣核酸(mRNA)密碼序列由不同tRNA攜帶相對應共價鍵結的胺基酸於核醣體接受醯化tRNA的A位佔位,接續依密碼合成全長胜肽鍊,直到接觸停止密碼,大腸桿菌的脫離因子1 (RF1)對TAG與TAA停止密碼並及脫離因子2 (RF2)分別與TGA及TAA停止密碼進行佔位並終止合成,全長胜肽鏈自核醣體脫離。
在上個世紀中期,已有研究顯示自然胺基酸的側鏈具有原子置換的類似物,如甲硫胺酸(methionine)的硫原子置換為硒原子,仍為甲硫胺醯轉核醣核酸合成酶(methionyl-tRNA synthetase, MetRS)的受質,同樣含氟的苯基丙胺酸(phenylalanine)也是苯基丙胺醯轉核醣核酸合成酶(phenylalanyl-tRNA synthetase, PheRS)的受質之一。這些發現都闡述著自然AARS的活性中心並非完美地只針對自然受質,因為人造類似胺基酸受質也並未出現於自然演化歷史中,所以在AARS提高正確性的除錯機制的演化上也並未遭遇過並避免;在適當的情況下,加入這些類似物也會使蛋白合成產生單一密碼組合有兩種胺基酸轉譯的情形,也造成了蛋白生合成的混合物。
在本世紀初,在生物正交基因工程的努力下,Methanocaldococcus jannaschii TyrRS•MjtRNACUA(圖一)在大腸桿菌中得到了證實1。在密碼子的選擇上,因為在大腸桿菌中TAG停止碼僅佔所有停止密碼的4%,因此再與RF1脫離因子競爭下,成功表現全長蛋白下,對原本細菌體的整體影響也相對較小,這也取決於細菌基因體在重要的基因結尾具有多個不同的停止密碼。接著在古生物菌發現且進一步在實驗室基因工程及導向演化的吡咯賴胺醯轉核醣核酸合成酶(pyrrolysyl-tRNA synthetase, PylRS)•tRNACUA2及磷酸絲胺醯轉核醣核酸合成酶(O-phosphoseryl-tRNA Synthetase, SepRS)•tRNACUA4皆達成生物正交。PylRS•tRNACUA配對的tRNA屬於天然形成的且具有CUA的反密碼子,這也讓科學家對於原生的古生物菌的蛋白質體大加研究,以瞭解這個非典型基因解碼方式的生命體,如何運作TAG停止碼為具意義的轉譯密碼(sense codon)。
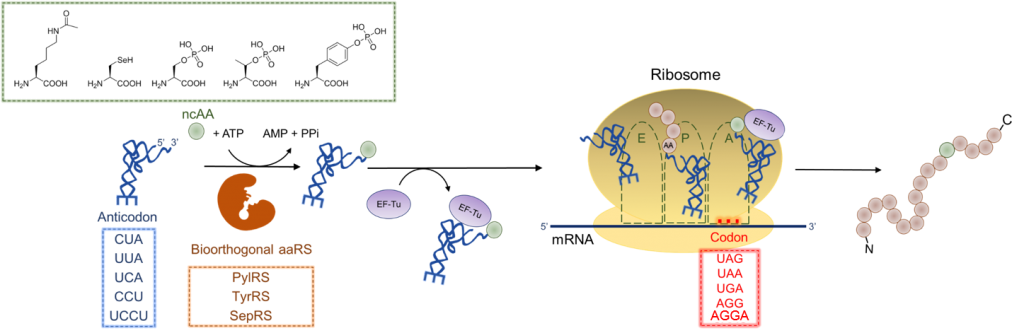
(圖一) 細菌非典型胺基酸體內嵌入系統。大腸桿菌中利用具有辨認停止密碼: TAG,TAA,及TGA;稀有密碼(rare codon): AGG及四鹼基密碼(four base codon)AGGA反密碼子(anticodon)的tRNA經由生物正交胺基醯化-轉核醣核酸合成酶 (biorthogonal aminoacyl-tRNA synthetase (aaRS))於體內嵌入非典型胺基酸於相對應信使核醣核酸(mRNA)位點,經 核醣體(ribosome)合成全長蛋白且具有位置特異性非典型胺基酸。圖中所左上角所列為目前已發展可以直接以自然核 醣體或增加工程後核醣體(針對AGGA四鹼基密碼子)生合成系統體內嵌入與蛋白轉譯後修飾有關的非典型胺基酸;由 左至右非典型胺基酸分別為N ε -acetyl-L-lysine (AcK),L-selenocysteine (Sec),phospho-L-serine (pSer),phospho- L-theorine (pThr)及phospho-L-tyrosine (pTyr)。 PylRS 由於和PheRS β domain 共享基
PylRS由於和PheRS b domain共享基因樹祖先(ancestor),在導向性的實驗室演化上,發現PylRS活性中心相對較大且容易形成對Phe類似物的非典型胺基酸,在受質廣度測試上發現具有多向特異性的受質活性中心(substrate poly-specificity),單就PylRS•tRNACUA配對,就已經發表超過兩百種非典型胺基酸體內嵌入的例子,其中對蛋白後轉譯修飾研究相當重要的AcK及pTyr體內嵌入(圖一),皆由此配對工程演化而得。PylRS•tRNACUA配對因為tRNA與PylRS相互作用並不涉及反密碼子區域的作用,因此tRNA反密碼子區域突變為解碼其他停止碼如UUA或CAU,稀有密碼CCU,甚至UGGU四密碼子(圖一),仍能維持PylRS活性及發展更多相互正交的PylRS•tRNACUA配對相當適合。唯四密碼子解碼需要在細菌中額外加入工程後的核醣體以準確解碼轉譯。PylRS•tRNACUA配對卓越之處是此配對在不同的模式生物皆展現正交性質,因此目前此配對可在酵母菌,哺乳動物細胞,果蠅,線蟲,斑馬魚,阿拉伯芥及小鼠等模式生物中操作2。此外,SepRS•tRNACUA配對在深入基因工程後的受質只有幾種,Sec, pSer及pThr這三個與蛋白後轉譯修飾密切相關的非典型胺基酸(圖一)卻是非常重要,且對於相關蛋白激酶(kinase)及硒蛋白(selenoprotein)研究提供重要工具及深遠影響。
在細菌基因體工程上,日益精進的合成生物學觀點及基因編輯技術,甚至是合成細菌基因體技術,將停止密碼賦予意義或縮減細菌基因體密碼使用種類。在賦予停止密碼意義上,RF1脫離因子基因剔除及調整為細菌正常生存基因下游停止密碼(圖二右上)5,做到了將停止碼轉變為轉譯密碼,同時也增進了單一蛋白可嵌入單一非典型胺基酸達30位點6,也更接近一般蛋白轉譯具多個位點的組合及蛋白設計的自由度。近幾年,細菌基因體全合成的嘗試,除了在基因編碼及基因體藍圖的交互考量下,除了TAG停止碼,基因密碼的簡併性(condon degeneracy),如六個密碼子簡併轉譯為亮胺酸(leucine)-TTA及TTG或絲胺酸(serine)-TCG及TCA,以及稀有密碼AGG及AGA。在577及618個縮減密碼子的細菌基因體建構(圖二下半部),目前61密碼子基因體證實可以維持類似的細菌生長及繁殖7。也為同時放入三個正交配對且在蛋白設計可運用23個胺基酸建立平台。在合成生物學的想像中,更有劃時代的科學進展,新的基因鹼基配對XY運用疏水性作用力(圖三),並與ATGC氫鍵配對相互正交9。由於外加XY去氧核醣核酸,體外運輸至細菌體內及半生期在測試後,可以對質體成功進行轉錄。在遽增的216 (6×6×6)個密碼子組合下,扣除細菌64個原始密碼子,156個新增密碼子,將MjTyrRS•MjtRNACUA 及PylRS•tRNACUA兩個生物正交配對的 tRNA反密碼子區域突變為含X或Y的反密碼子,成功的在大腸桿菌中完成含XY的編碼轉譯並嵌入非典型胺基酸與設計位點10。這樣的研究結果也預示足夠的密碼子以容納更多正交的AARS•tRNA配對。
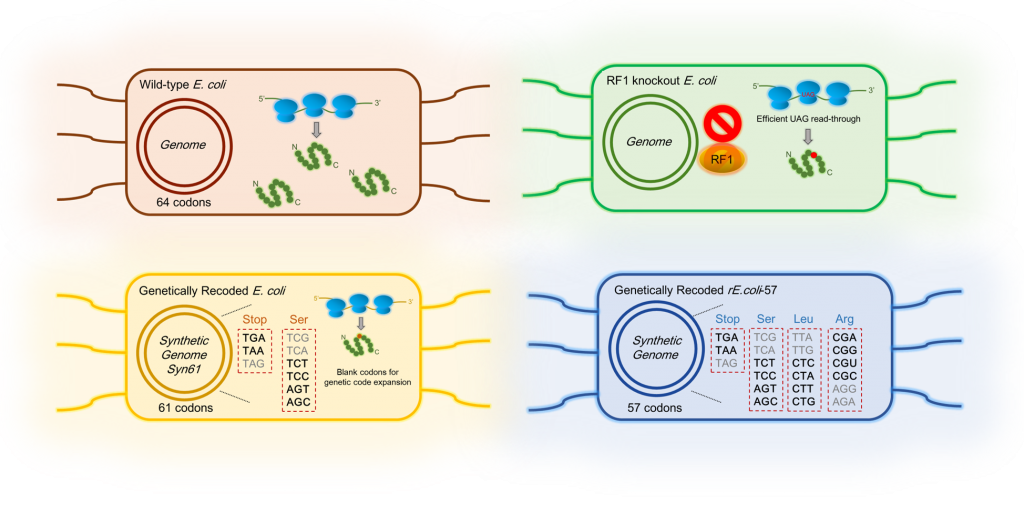
(圖二) 野生株大腸桿菌(E. Coli )經過基因工程及新型基因合成技術,減少基因密碼使用數量,作為新型AARS•tRNA配對 使用。左上為野生株大腸桿菌,正常使用61組轉譯基因密碼及3組停止密碼(TAG,TAA及TGA)。右上為將RF1蛋白基 因自大腸桿菌基因體中剔除,導致TAG停止密碼被賦予為轉譯基因密碼。以右上△RF大腸桿菌基因體為藍本,利用前 衛的基因合成方法分別剔除基因體中分別為TCG及TCG(左下)與TCG、TCA、TTA、TTG、AGG及AGA(右下),進一步合成 僅用61和57個基因密碼子的大腸桿菌。
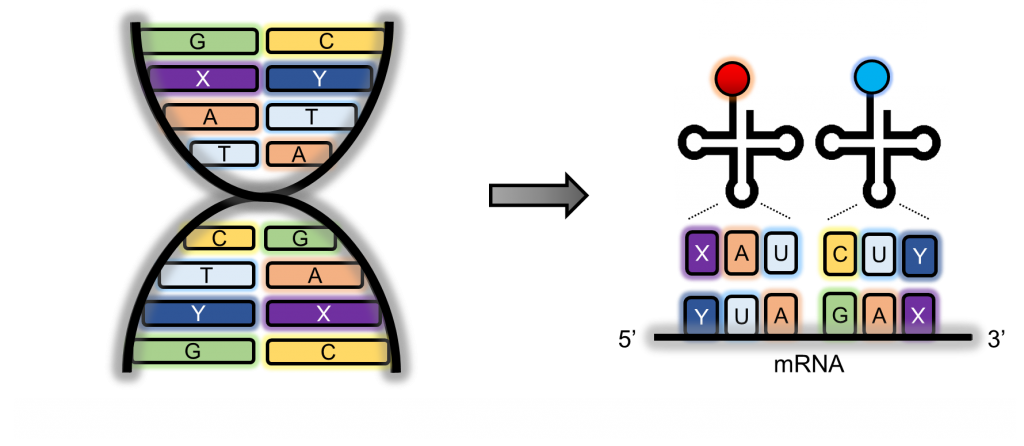
(圖三) 新型XY鹼基配對(base pair)經由大腸桿菌體內轉錄和轉譯嵌入非典型胺基酸。X及Y鹼基配對利用生物正交的疏水性 作用力(hydrophobic interactions),以及自然轉譯及轉錄的相容性下,具有更多樣基因密碼ATGCXY(63 = 216),完成 生產蛋白且展現更多樣性的非典型胺基酸嵌入潛力
藉由開闢的細菌密碼子縮減基因體或創造新的正交基因密碼XY,皆提供數個到156個新的基因編碼提供新型AARS•tRNA配對轉譯解碼以嵌入多種非典型胺基酸。目前該領域發展以PylRS•tRNACUA中無N端區段的PylRS以及工程tRNA以維持相互正交,並成功以不同停止密碼TAG及TAA分別具位置特異性嵌入兩種不同非典型胺基酸。這個領域未來的研究應著重於更多相互正交的AARS•tRNA的建立,以及優化AARS的催化效率以勝任於單一蛋白中多位點非典型胺基酸嵌入,進一步應用於新穎蛋白設計及功能探討。
參考文獻
- Liu, C. C. and Schultz, P. G., Adding new chemistries to the genetic code., Rev. Biochem. 79 (2010) 413-44.
- Chin, J. W., Expanding and reprogramming the genetic code. Nature, 550 (2017) 53-60.
- O’Donoghue, P., Ling, J., Wang, Y. S. and Söll, D. Upgrading protein synthesis for synthetic biology. Chem. Biol. 9, (2013), 594-598.
- Park, H. S. et al., Expanding the genetic code of Escherichia coli with phosphoserine., Science 333 (2011), 1151-4.
- Mukai, T., et al. Codon reassignment in the Escherichia coli genetic code. Nucleic Acids Res.38 (2010) 8188-8195.
- Amiram, M., et al, Evolution of translation machinery in recoded bacteria enables multi-site incorporation of nonstandard amino acids, Biotechnol. 33 (2015) 1272–1279.
- Fredens, J., et al. Total synthesis of Escherichia coli with a recoded genome. Nature569 (2019) 514-518.
- Ostrov, N., et al. Design, synthesis, and testing toward a 57-codon genome. Science353 (2016) 819-822.
- Malyshev, D. A. et al., A semi-synthetic organism with an expanded genetic alphabet, Nature 509 (2014) 385-388.
- Zhang, Y. et al, A semi-synthetic organism that stores and retrieves increased genetic information, Nature 551 (2017) 644–647.
